Микроорганизмы рода Lactobacillus обладают антагонистической активностью в отношении патогенных микроорганизмов и выполняют иммуномодулирующую функцию. Положительное влияние лактобацилл на здоровье человека объясняет их активное использование в пробиотиках. Актуальным вопросом является получение новых данных о биологических свойствах и молекулярно-генетической структуре лактобацилл, создание новых пробиотических препаратов на их основе с помощью модифицированных подходов к культивированию. Результаты определения видов L. acidophilus L. casei, L. Rhamnosus, L. paracasei, полученные классическим биохимическим методом идентификации на основе сахаролитической активности, затрудняющие проведение видовой идентификации, сопоставимы с молекулярно-генетическим методом. В случае с L. casei, L. rhamnosus, L. paracasei, данные полученные с использованием ПЦР, позволяют уточнить таксономическое положение исследуемых штаммов.
Ключевые слова: микроорганизмы рода Lactobacillus, пробиотики, выделение ДНК, биохимическия идентификация, ПЦР.
Микроорганизмы рода Lactobacillus выполняют иммуномодулирующую функцию и обладают антагонистической активностью в отношении патогенных микроорганизмов. Положительное влияние лактобацилл на здоровье человека объясняют их активное использование в пробиотиках. Позитивные эффекты нормальной микрофлоры кишечника и пробиотиков обусловлены в основном бифидо- и лактобактериями.
Угнетение роста нежелательных микробов происходит благодаря продукции пробиотиками субстанций с выраженной антагонистической активностью: лизоцима, бактериоцинов, органических кислот (молочной, уксусной, янтарной, муравьиной), перекиси водорода, веществ с антибиотической активностью. Образование лактобациллами молочной кислоты приводит к снижению pH внутрикишечного содержимого до pH 4,0–5,8 и сдерживанию роста и размножения гнилостных микроорганизмов. Пробиотики обладают адгезивной активностью к эпителиальным клеткам кишечника и конкурируют с патогенными и условно патогенными микробами за сайты адгезии на кишечной стенке, что в итоге также ведет к угнетению роста нежелательной микрофлоры [1].
Группа молочнокислых бактерий включает в себя микроорганизмы филогенетически близких родов: Lactococcus, Lactobacillus, Streptococcus, Leuconostoc, Enterococcus, Oenococcus, Carnobacterium, Weissella, Alloicoccus, Dolosigranulum, Melissicoccus, Tetragenococcus, Vagococcus, Lactosphaera, Aerococcus.
В пробиотикотерапии используются виды Lactobacillus: L. acidophilus, L. rhamnosus, L. plantarum, L. fermentum, L. delbrueckii subsp. lactis, L. delbrueckii subsp. bulgaricus, L. reuteri, L. casei [2].
Для совместного культивирования считают биосовместимые штаммы, основанные на симбиотических взаимоотношениях (симбиоз, метабиоз, сателлитизм, синергизм). В кефирных зернах одновременно развиваются молочнокислые бактерии и дрожжи, при этом молочнокислые бактерии, испытывающие потребность в витаминах, получают их в результате развития дрожжей, последние получают благоприятные условия для развития за счет подкисления среды. Разновидностью метабиоза является сателлитизм, для которого характерно, что одни микроорганизмы выделяют в среду ростовые вещества (аминокислоты, витамины и др.), стимулирующие развитие другого микроорганизма. При синергизме у членов микробной ассоциации взаимно повышается физиологическая активность за счет выделения продуктов, стимулирующих их развитие.
Актуальным вопросом является получение новых знаний о биологических свойствах и молекулярно-генетической структуре лактобацилл; создание новых пробиотических препаратов на их основе.
Критерием межродовой и видовой идентификации Lactobacillus являются биохимические и морфологические свойства. Альтернативой классической биохимической идентификации ученые предлагают метод генотипирования с использованием полимеразной цепной реакции (ПЦР).
ПЦР применяется при подборе штаммов-продуцентов на этапе идентификации и для контроля штаммового состава на всех этапах производства пробиотических препаратов.
Внедрение ПЦР в процесс контроля пробиотических препаратов в производственном процессе позволит перейти на новый методический уровень обеспечения качества и эффективности многокомпонентных препаратов-пробиотиков [3].
Метод ПЦР широко применяется исследователями для работы с микроорганизмами p. Lactobacillus, Streptococcus, Bifidobacterium.
Родовая и видовая идентификации бактерий рода Lactobacillus позволяет усовершенствовать контроль технологического процесса на предприятиях по производству фармакопейных препаратов, БАД к пище и продуктов питания на основе молочнокислых микроорганизмов и ставит на новый уровень контроль мультиштаммовых пробиотических продуктов.
Для дизайна родо- и видоспецифичных праймеров используют генетические детерминанты 16S и 23S рРНК.
Для работы с микроорганизмами молочнокислого брожения используются варианты ПЦР — nested-ПЦР (гнездовая ПЦР) и мультиплексная ПЦР [4]. В мультиплексных ПЦР системах для выявления каждого возбудителя, как правило, используется своя пара праймеров. При работе с лактобациллами и бифидобактериями чаще всего используется вариант мультиплексной ПЦР, при котором общим (родоспецифичным) является праймер-реверс, а праймеры-форварды — видоспецифичны [5].
Для субтипирования бактерий рода Lactobacillus используют метод RAPD (Random Amplification of Polymorphic DNA), ПЦР со случайной амплификацией полиморфной ДНК — когда нужно различить сходные геномы (виды бактерий). В этом методе обычно используют небольшой праймер (до 10 п.н.), который может гибридизоваться со многими случайными участками генома.
Амплификация фрагмента гена 16S рРНК для индикации и видовой идентификации пробиотических штаммов лактобацилл сопоставима с классической биохимической идентификацией.
Материалы и методы, использованные в исследованной работе .
В работе использовали коммерческий лиофильно высушенные препараты Линекс, Бифидобактерин бифидум, кисломолочный продукт Нарине.
Культуры бактерий рода Lactobacillus выращивали на среде MRS-4, инкубация — 12 ч. при температуре 37 °С.
Для культивирования бактерий рода Lactobacillus использована среда MRS (de Man, Rogossa and Sharpe), богатая питательными, веществами и ростовыми факторами. Среда содержит декстрозу, бактериологический пептон, мясной экстракт, ацетат натрия, дрожжевой экстракт, гидроортофосфат калия, цитрат аммония, Твин 80, сульфат магния, сульфат марганца, бактериологический агар, конечная величина pH 6,2 ± 0,2 при 25 °C [4].
Биохимическая идентификация бактерий рода Lactobacillus (в процессе определения ферментации углеводов) проведена с использованием стандартных биохимических тест-стрипов RapID. Интерпретация результатов проводится программным обеспечением ERIC.
В состав пестрого ряда набора стандартных биохимических тестов-стрипов RapID входят субстраты сахаров и многоатомных спиртов: арабиноза, целлобиоза, галактоза, лактоза, мальтоза, маннит, манноза, мелибиоза, раффиноза, салицин, сахароза, трегалоза, ксилоза, сорбит.
После проверки чистоты культуры с помощью микроскопии, с поверхности агаровой пластины снимают выросшие колонии петлей и ресуспендируют в стерильном растворе хлорида натрия для получения взвеси, содержащей 1 миллиард клеток бактерий в 1 мл по стандарту мутности. Полученную взвесь бактерий в количестве 0,01 мл вносят в 1 мл основы (концентрация взвеси клеток в основе среды — 10 7 м.к.).
Лиофильно высушенные штаммы разводят в 1 мл стерильного физиологического раствора и 0,1–0,15 мл и высевают на жидкую среду MRS-2. Инкубация — 2 суток, t — 37 °С. Разведения — 10– 1 –10– 6 в стерильном физиологическом растворе. Повторный высев на среду MRS-4. Инкубация — 2 суток, t — 37 °С. Через 2 суток культуры используют для постановки пестрого ряда на биохимических тест-стрипах. Инкубация — 4 часа (аэробно), t — 37 °С. Результаты учитывают по изменению окраски среды в лунках.
Для выделения геномной ДНК бактерий рода Lactobacillus, используют метод лизиса клеток с помощью фенола, хлороформа или хаотропным агентом — гуанидин тиоционатом; центрифугирование для удаления денатурированных белков и фрагментов клеточных органелл и последующей сорбцией ДНК на носителе (метод нуклеосорбции). После отмывок в пробе остается ДНК, сорбированная на носителе SiO 2 , с которого она выводится элюирующим буфером. Метод нуклеосорбции удобен, технологичен и пригоден для подготовки образца к амплификации [5].
ДНК выделяется из 12-ти часовой культуры при оптической плотности бактериальной взвеси 10 9 микробных клеток в 1мл раствора. 1 мл раствора центрифугируют (3000 об/мин,1 мин); 100 мкл центрифугата используется для выделения ДНК. Сухие, лиофильно высушенные препараты разводят в 1 мл стерильной дистиллированной воды; 100 мкл суспензии используют для выделения ДНК.
Состав набора «ДНК-сорб-АМ»: лизирующий раствор, отмывочный раствор, универсальный сорбент, ТЕ-буфер для элюции.
Лизис 100 мкл культуры — 300 мкл буфера (6М гуанидинтиоцианат, 0,01M ЭДТА, 20 мМ дитиотрейтол); инкубация — 65 °С, 10 мин. Нуклеосорбция — 25 мкл SiO 2 ; осаждение сорбента — центрифугирование (5000 об/мин, 30 с); отмывка сорбента — 4M раствор GuSCN х 1, буфером с этиловым спиртом (10 мМ Tris-HCl, 50 мМ NaCl, 50 % этанол) х 2. Высушивание осадка — 65 °С, 10 мин. Элюирование ДНК — 80 мкл буфера ТЕ (10мМ Tris HCl, 1мМ EDTA, рН — 8,0). Результаты исследования учитываются путем анализа исследуемых образцов методом электрофореза в 1,5 % агарозном геле.
Молекулярно-генетическая идентификация штаммов молочнокислых бактерий базируется на определении последовательностей информационного гена 16S рРНК.
Реакционная смесь для постановки ПЦР: 10 мкл ДНК, 2 мкл ΣdNTP, 2 мкл буфера ТЕ, по 1 мкл прямого и обратного праймеров, 10 мкл ПЦР-смеси-2.
Программа амплификации: денатурация геномной ДНК — 94 °С, 5 мин; отжиг праймеров — 55 °С, 30 сек; элонгация — 72 °С, 45 сек. После 35 циклов амплификации — заключительная элонгация — 72 °С, 3 мин. Хранение при 4 °С [6]. Амплификатор типа «Терцик» (ДНК-технологии, Москва).
Для индикации бактерий рода Lactobacillus использованы праймеры специфичные в отношении гена 16S рРНК. В исследованиях молекулярно-генетических исследованиях для индикации бактерий рода Lactobacillus использованы родоспецифичные праймеры casei/Y2, rham/Y2, para/Y2, RAPD (Ward L.) [7]. Олигонуклеотидные праймеры синтезированы в ООО «СибЭнзим», г. Новосибирск.
Молекулярно-генетический анализ таксономической принадлежности бифидобактерий.
Реакционная смесь для постановки ПЦР (на 100 мкл): 10 мкл 10×ПЦР буфера, 10 мкл смеси 2,5 mM ΣdNTPs, 4 мкл 50 mМ MgCl 2 , 300 нг геномной ДНК, 0,8 мкл фермента Tag-полимеразы. Олигонуклеотидные праймеры добавляют в концентрации 20 пмоль на 100 мкл смеси.
Программа амплификации: денатурация геномной ДНК при 95 °С, 5 мин; 30 циклов амплификации: 94 °С в течение 1 мин (денатурация), 60 °С, 1 мин (отжиг олигонуклеотидов), 72 °С, 2 мин (элонгацияцепи); финальная элонгация фрагментов — 72 °С, 10 мин. Хранение при 4 °С.
Для индикации рода и вида бифидобактерий будут использованы праймеры g-Bifid 1,2; BiBIF-1,2 (соответственно):
g-Bifid-1 5'-CTCCTGGAAACGGGTGG-3'
g-Bifid-2 3'-GGTGTTCTTCCCGATATCTACA-5'
Ожидаемый размер ПЦР фрагмента, нуклеотиды — 549–563 п.н.
BiBIF-1 5'-CCACATGATCGCATGTGATTG-3'
BiBIF-2 5'-CCGAAGGCTTGCTCCCAAA-3'
Ожидаемый размер ПЦР фрагмента, нуклеотиды — 278 п.н.
Ампликоны разделяются электрофорезом в агарозном геле и визуализируются после окраски бромидом этидия (камера для горизонтального электрофореза «SE-2»).
Электрофорез продуктов амплификации — 1,5–2 % агарозный гель, 5 мкг/мл бромистого этидия; трис-боратный буферный раствор (0,089 M трис, 0,002 M ЭДТА, рН–8,0). К пробе добавляют 2,5 мкл буфера (электродный буфер, 50 % глицерин и 0,1 % бромфеноловый синий), время разгонки — 40 мин.
Визуализация результатов — УФ свет, длина волны 254 нм (трансиллюминатор TCP-20 МС). Размер получаемых фрагментов ДНК определяют путем сравнения-электрофоретической подвижности с фрагментами ДНК маркера (100–1000 п.н.).
Для изучения антагонистических взаимоотношений лактобацилл использован метод, основанный на одновременном совместном культивировании испытуемых штаммов. Через определенные промежутки времени отбирают пробы по 1 мл всех культур и после соответствующих разведений по 0,1 мл высевают на селективные для каждого штамма среды. Чашки помещают в термостат и после инкубирования подсчитывают количество сформировавшихся колоний. Чистоту культуры клеток проверяют путем повторного рассева на селективные среды, обеспечивающие избирательный рост тех или иных микроорганизмов. Критерием чистоты в этом случае является однородность формирующихся при этом колоний.
Результаты работы.
Основа видовой идентификации лактобацилл — биохимические свойства лактобацилл по способности ферментировать углеводы. Классическая микробиологическая схема идентификации основана на метаболизме сахаров. Субстраты утилизируются гетероферментативными и гомоферментативными лактобациллами по гликолитическому и окислительному пентозофосфатному пути.
В работе представлены данные биохимических свойств видов бактерий рода Lactobacillus — L. acidophilus, L. сasei., L. rhamnosus (таблица 1). Биохимические свойства штаммов бактерий рода Lactobacillus определены с применением тест-систем RapID.
Биохимические свойства видов L. casei, L. rhamnosus, L. paracasei сходны, что затрудняет проведение видовой идентификации. Наблюдаются одинаковые реакции ассимиляции арабинозы, целлобиозы, галактозы, лактозы, мальтозы, маннита, маннозы, мелибиозы, раффинозы, салицина, сахарозы, трегалозы, ксилозы, сорбита и т. д. перечисленными лактобациллами. По данным источников, L. rhamnosus — это бактерия, которая первоначально считалась подвидом L. casei, но генетические исследования показали, что она является отдельным видом. Видовая дифференциация B. bifidum, L. acidophilus прослеживается.
Альтернативным методом определения родовой и видовой идентификации бактерий рода Lactobacillus являются молекулярно-генетические характеристики.
Перед выделением ДНК нуклеосорбционным методом, Штаммы бактерий лактобацилл и изоляты кисломолчного продукта (ТОО «Компания ФудМастер Трэйд»), исследованные в работе, культивировали в бульоне MRS (De Man et al.) аэробно при температуре 30 °C.
Таблица 1
Биохимические свойства бактерий рода Lactobacillus
|
Виды |
Целлобиоза |
Галактоза |
Лактоза |
Мальтоза |
Маннит |
Манноза |
Мелибиоза |
Раффиноза |
Салицин |
Сахароза |
Трегалоза |
Арабиноза |
Сорбит |
Ксилоза |
Эскулин |
|
B. bifidum |
± |
+ |
+ |
+ |
± |
+ |
+ |
+ |
- |
+ |
± |
- |
- |
- |
+ |
|
L.acidophilus |
+ |
+ |
+ |
+ |
- |
+ |
- |
+ |
+ |
+ |
- |
- |
- |
- |
+ |
|
L. casei |
+ |
+ |
+ |
+ |
+ |
+ |
- |
- |
+ |
+ |
+ |
- |
+ |
- |
+ |
|
L. rhamnosus |
+ |
+ |
+ |
+ |
+ |
+ |
- |
- |
+ |
+ |
+ |
- |
+ |
- |
+ |
|
L. paracasei |
+ |
+ |
+ |
+ |
+ |
+ |
- |
- |
+ |
+ |
+ |
- |
+ |
- |
+ |
|
Примечание: «+» положительный; «-» отрицательный; «±» — замедленно положительный | |||||||||||||||
Амплификацию ДНК из гена 16S рРНК проводят при следующих условиях амплификации: 94 °C 3 мин; 45 °C 45 сек; 72 °C 1 мин; 30 циклов. Продукты амплификации разделяют на 2 % агарозном геле.
Для видового типирования Lactobacillus методом ПЦР используется ген 16S рРНК, несущий консервативные и вариабельные участки нуклеотидной последовательности. В работе планируется использовать праймеры ( Ward L. ):
– casei/Y2 5'-TGCACTGAGATTCGACTTAA-3' Lact. casei 16S;
– rham/Y2 5'TGCATCTTGATTTAATTTTG-3'Lact. rhamnosus 16S;
– para/Y2 5'-CACCGAGATTCAACATGG-3'Lact. paracasei 16S;
– RP 5' - CAGCACCCAC - 5' RAPD primer.
Праймер RAPD используется при идентификации видов Lactobacillus по наличию специфических амплификационных фрагментов для сравнения.
При гибридизации праймеров casei/Y2, rham/Y2, para/Y2 с 16S рРНК L. casei, L. rhamnosus, L. paracasei амплифицированы фрагменты примерно 290 п.н. от каждого из штаммов.
При ПЦР с RAPD праймером (RP) просмотрены специфические амплификационные фрагменты штаммов L. paracasei, L. rhamnosus. Дифференцированы близкородственные виды L. paracasei и L. rhamnosus; размеры полученных ампликонов — 850, 550 п.н., соответственно.
Ампликоны, идентифицированные как L. paracasei, содержат фрагмент RAPD 850 п.н. (полосы 2–10), а штаммы L. rhamnosus содержат фрагмент RAPD 550 п.н. (полосы 1–10) (рис. 1. c, d)
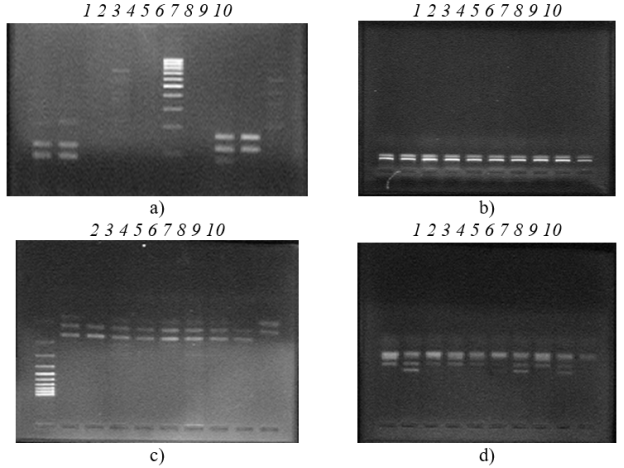
Рис. 1. Электрофореграмма результата ПЦР генов Lactobacillus: a)1 — L. casei (casei/Y2, 290 п.н.); 2 — L. paracasei (casei/Y2, 290 п.н.); 8, 9 — L. rhamnosus (rham/Y2, 290 п.н.); b) 1–10 L. rhamnosus (rham/Y2, 290 п.н.); c) 2–10 L. paracasei (RP 850 п.н.); d) 1–10 L. rhamnosus (RD 550 п.н.)
Результаты идентификации видов L. acidophilus L. casei, L. rhamnosus, L. paracasei полученные классическим биохимическим методом идентификации на основе сахаролитической активности сопоставим с ПЦР. В случае с L. casei, L. rhamnosus, L. paracasei, данные, полученные с использованием ПЦР, позволяют уточнить таксономическое положение исследуемых штаммов. Молекулярно-генетический метод является ценным дополнением для идентификации лактобацилл, учитывая вариабельность классического биохимического метода.
При создании мультипробиотиков необходимо изучению межштаммовых взаимодействий лактобацилл при их совместном культивировании.
В качестве индикаторных штаммов использовались препараты пробиотиков «Lactobacillus acidophilus LA-5» производства Chr. Hansen, Lactobacillus casei DN-114 001(Actimel, DanActive) производства Danone/Dannon, Lactobacillus rhamnosus ATCC 53013 (LGG) производства Valio. В состав лактобактерина Lactobacillus acidophilus LA-5 входят Lactobacillus acidophilus LA-5, в количестве не менее 2×10 9– 4×10 9 м. к. Состав Actimel представлен Lactobacillus casei DN-114 001, Lactobacillus rhamnosus ATCC 53013 (LGG) производства Valio в количестве не менее 10 7 м. к. в одной дозе.
Для изучения антагонистической активности использовали метод прямого антагонизма (смешанной культуры ) . 1,0 мл изучаемого штамм-продуцент (разведение 10 7 ) вносили в 10,0 мл полужидкой среды МРС-4. Индикаторные культуры вносили в количестве 0,1 мл от 10 7 . Суточную культуру, выращенную на жидкой питательной среде, наносили на поверхность плотной питательной среды. Для обнаружения и количественного учета Lactobacillus высевали (0,1 мл суспензии) через 12 ч, 24 ч на плотную питательную среду МРС [8].
При высеве образцов из смешанной культуры через 12 ч наблюдали хороший рост в 10 8 –10 9 КОЕ/мл всех лактобактерий. В течение суток во всех пробах штаммы L. rhamnosus продолжали расти в опытных пробирках, чего не наблюдалось в случае L. acidophilus и L. casei. После 24 часов инкубации смесей наблюдали L. acidophilus и L. casei в концентрации не менее 10 6 КОЕ/мл.
Взаимоотношение пробиотических штаммов бактерий рода Lactobacillus показали, что биосовместимыми являются штаммы L. acidophilus L. casei. Штамм L. rhamnosus проявляет сильную антагонистическую активность, действует подавляюще на штаммы L.acidophilus L. casei.
Литература:
- Ермоленко Е. И. Определение антагонистической активности лактобактерий / Е. И. Ермоленко, А. Н. Суворов, A. B. Воейкова // Пробиотики, пребиотики, синбиотики и функциональные продукты питания. Современное состояние и перспективы: Сб. матер. Междунар. Конф. 2–4 июня. — Москва, 2004. — С. 25–26.
- Бондаренко В. М. Классификация бактерий рода Lactobacillus / В. М. Бондаренко // Матер.VІІІ съезда Всеросс. Общества эпидемиол., микробиол. и паразитол. — М., 2002. — Т. І. — С. 140.
- Комбарова С. Ю. Молекулярно-генетические методы дифференциации промышленных штаммов бифидобактерий и лактобацилл / С. Ю. Комбарова, Т. С. Погосьян, 0. Ю. Борисова и др. // Сборник материалов конференции «Пробиотические микроорганизмы — современное состояние вопроса и перспективы использования» — М., 2002. — С. 21.
- Беспоместных К. В. Изучение влияния состава питательной среды на изменение биохимических и морфологических свойств штаммов лактобацилл // Современные проблемы науки и образования. — 2014. — №
- Moore D. Purification and Concentration of DNA from Aqueous Solutions // Current Protocols in Pharmacology — 2007. — Vol. 38, № 1 — Р. 1– 10
- Kwon H. S. Rapid identification of probiotic Lactobacillus species by multiplex PCR using species-specific primers based on the region extending from 16S rRNA through 23S rRNA / H. S. Kwon, E. H. Yang, S. W. Yeon et al // FEMS Microbiology Letters. — 2004. — Vol. 239, Is.2. — P. 267–275.
- Ward L. J. Differentiation-of Lactobacillus casei, Lactobacillus paracasei and Lactobacillus, rhamnosus by polymerase chain reaction / L. J. Ward, M. J. Timmins // Lett Appl Microbiol. — 1999: — Vol. 29. — P. 90–92.
- Bhatia S. J., Kochaz N., Abraham P. et al. J. Clin. Microbiol. — 1989. — V. 27. — P. 2328–2330.

