Ключевые слова: машинное зрение, сверточная нейронная сеть, МРТ, глубинное обучение.
С каждым днем потребность в технологии машинного обучения постоянно возрастает. С ее помощью стало возможным решить множество задач, связанных с распознаванием речи и текста, классификацией и сегментацией объектов на изображениях и т. д. Совершенно естественно, что данная технология нашла свое применение и в медицине. Например, сегодня существует проблема с диагностированием у пациентов опухолей головного мозга: нередки такие ситуации, когда определение новообразований происходит на поздней стадии их развития, при которой лечение человека практически невозможно.
Магнитно-резонансная томография (МРТ) является одной из самых прорывных технологий диагностики опухолей: она получает послойные изображения тканей мозга с разных ракурсов с толщиной слоя до 8 мм, что позволяет довольно точно локализовать новообразования еще на ранних стадиях развития [1]. Несмотря на то, что МРТ была изобретена еще в 70-х годах прошлого столетия, на сегодняшний день она все еще является наиболее популярным методом диагностики заболеваний головного мозга (Альцгеймер, эпилепсия, рак и т. д.). Но анализ МРТ снимков вручную является довольно долгим и неэффективным методом из-за быстрой утомляемости людей от рутинной работы и, вследствие этого, человеческих ошибок [2]. Соответственно, если компьютер научится быстро и точно распознавать опухоль головного мозга на ранних стадиях развития по МРТ снимкам, то удастся значительно уменьшить число врачебных ошибок, повысить вероятность выявления данной болезни на ранних стадиях развития у большего числа пациентов и тем самым спасти множество жизней. В дополнение ко всему этому, автоматизация рутинной работы по анализу МРТ снимков поможет разгрузить врачей.
Существует множество различных типов злокачественных образований головного мозга. Наиболее распространенной и агрессивной формой опухолей головного мозга является глиобластома. Обнаружение и локализация таких образований производятся по послойным снимкам МРТ головного мозга. Съемка головного мозга производится в нескольких режимах работы томографа. Самый распространенный режим — Т1 ВИ. В данном режиме белым цветом обозначается жир, мягкие ткани отображаются оттенкам серого, а спинномозговая жидкость — черным. Так же во время съемки, пациенту может быть введено особое вещество — контраст (режим T1c ВИ). На снимках, полученных с применением контрастного вещества, пораженные участки подсвечиваются ярким белым светом, сильно выделяясь на фоне мягких тканей. Для локализации жидкостей используют режим Т2. В данном режиме жидкости и жир отмечаются белым цветом, что позволяет засечь перифокальный отек, образующийся вокруг опухоли. Для того чтобы отличить перифокальный отек от других жидкостей головного мозга используется режим FLAIR.
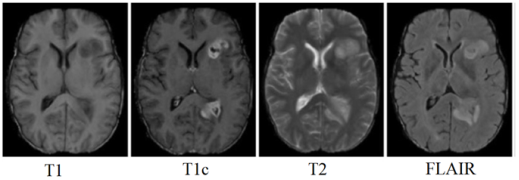
Рис. 1. Снимки МРТ головного мозга в различных режимах [3]
В области распознавания образов на изображениях, наибольших успехов достигли сверточные нейронные сети, получившие такое название за счет использования сверточных слоев. Принцип работы сверточного слоя довольно прост: по входному многомерному массиву данных двигается матрица весов, называемая фильтром. Область данных, на которую применяется фильтр, называется его полем восприятия. При перемещении фильтра происходит поэлементное перемножение матрицы на элементы поля восприятия. Затем сумма полученных значений записывается в матрицу выходных значений, называемой картой признаков.
Рис. 2. Визуализация свертки фильтром 5 х 5 и составление карты признаков [4]
Каждый из фильтров ищет какой-либо признак объекта распознавания (например, какой-нибудь цвет, простые линии и т. д.). Нетрудно догадаться, что чем больше фильтров мы используем, тем точнее получится результат распознавания. В итоге выходной массив получается меньше, чем исходный. Важно отметить, что в нейронной сети используется не один, а несколько сверточных слоев. Это позволяет ей распознавать сложные объекты. Вот как это работает: Карты признаков, полученная первым сверточным слоем и содержащая простые признаки, обрабатывается следующим слоем свертки, фильтры которой распознают сочетания простых элементов (например, прямоугольники, которые есть сочетание прямых линий). Последующие слои работают со все более замысловатыми формами распознаваемого объекта.
Между сверточными слоями ставятся слои с функцией активации и пулинга (Pooling). Самой популярной функцией активации является ReLU:
f(x) = max(0,x)(1)
Она позволяет не учитывать в расчетах нейроны, выходной сигнал которых отрицательный. Слой пулинга требуется для уплотнения нескольких соседних значений до одного (например, несколько пикселей преобразуются в один), что позволяет ускорить обучение сети и избежать её переобучения (запоминание сетью мельчайших свойств объекта, в результате чего она способна распознавать их только на обучающей выборке).
В настоящее время сверточные нейронные сети широко применяют в задачах распознавания опухолей на снимках МРТ головного мозга. В данной работе для этих целей мы использовали архитектуру UNet. В её основе лежит применение сжимающего и разжимающего путей сверточных слоев.
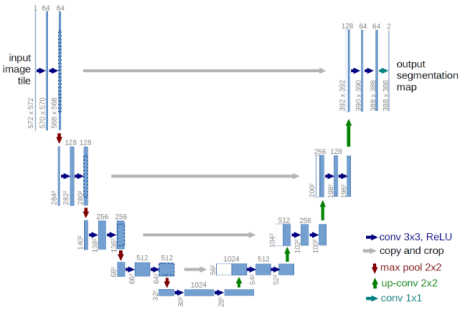
Рис. 3. Архитектура UNet [5]
Сжимающий путь основан на классической схеме нейронных сетей (свертка => ReLU => пулинг => свертка => ReLU => пулинг => …). Последний слой использует фильтры 1х1, чтоб сопоставить карту абстрактных признаков с вектором классов (в нашем случае классами будут являться отек вокруг опухоли, центр опухоли и опухолевые клетки в центре). Разжимающий использует слои обратной свертки и слои апскейлинга (Upscaling) (операция апскейлинга обратна пулингу). В ходе обратной свертки, апскейлинга, а также объединения карт признаков с картой, полученной на соответствующем шаге свертки (соединение необходимо для восполнения потери пограничных значений на каждой свертке), получается карта признаков с уточненными областями их расположения относительно первоначальных данных.
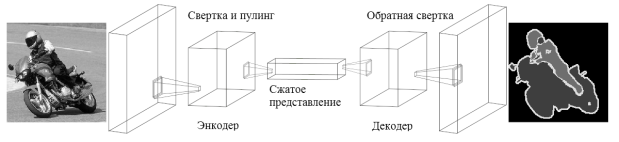
Рис. 4. Схема работы UNet [6]
Для обучения нейронной сети UNet использовался архив BRATS 2017 [7,8]. Данный архив содержит снимки МРТ головного мозга пациентов в формате nii.gz, снятых в режимах T1, T1c, T2 и FLAIR. Созданная программа использует фреймворк глубинного обучения TensorFlow и технологию построения нейронных сетей для распознавания медицинских изображений NiftyNet.
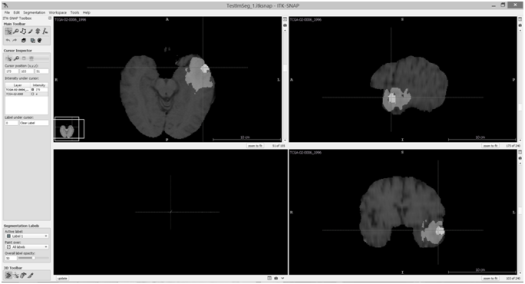
Рис 5. Результат работы нейронной сети: снимок МРТ головного мозга с отмеченными областями отека (светло-серая зона), центра опухоли (белая зона) и живых опухолевых клеток (темно-серая зона)
Результат работы программы записывается в виде файла формата nii.gz.
Для его просмотра необходимо открыть в программе просмотра медицинских изображений файл, загружавшийся в программу, и файл, содержащий результат работы. Мы использовали программу ITK-SNAP 3.6.0.
В итоге реализованная программа при обработке снимка МРТ головного мозга пациента формата nii.gz выдает результат, который отображается программой ITK-SNAP в виде изображение его мозга в трех проекциях (аксиальном, сагиттальном и фронтальном) с подсвеченными областями отека, центра опухоли и живых опухолевых клеток.
Литература:
1. Kullberg J. Whole-body adipose tissue analysis: comparison of MRI, CT and dual energy X-ray absorptiometry / Kullberg J. [и др.] // The British Journal of Radiology. –2014. — Т. 82. — № 974. — С. 123–130.
2. Akkus Z. Deep Learning for Brain MRI Segmentation: State of the Art and Future Directions / Akkus Z. [и др.] // Journal of Digital Imaging. — 2017. — Т. 30. — № 4. — С. 449–459.
3. Pereira S. Brain tumor segmentation using convolutional neural networks in MRI images / Pereira S. [и др.] // IEEE transactions on medical imaging. — 2016. — Т. 35. — № 5. — С. 1240–1251.
4. Nielsen, M. Neural Networks and Deep Learning / M. Nielsen. — San Francisco: Determination Press, 2015. — 224 c.
5. Румянцев В. Ф. Использование методов машинного обучения для семантической классификации дорожной обстановки: вкр. / В. Ф. Румянцев. — Санкт-Петербург, — 2018. — 23 с.
6. Silva, T. Deeplab Image Semantic Segmentation Network [Электронный ресурс]. / T. Silva // Thalles' Blog: — Режим доступа: https://sthalles.github.io/deep_segmentation_network (дата обращения: 27.05.2019).
7. Menze, BH. The Multimodal Brain Tumor Image Segmentation Benchmark (BRATS), / BH. Menze [и др.] // IEEE Transactions on Medical Imaging. — 2015. — Т. 34. — № 10. — С. 1993–2024. — DOI: 10.7937/K9/TCIA.2017.KLXWJJ1Q.
8. Bakas, S. Advancing The Cancer Genome Atlas glioma MRI collections with expert segmentation labels and radiomic features. / S. Bakas [и др.] // Nature Scientific Data. — 2017. — DOI: 10.1038/sdata.2017.117.

